研究内容
1.水圏生物における分子生態学的研究
1)DNA分析による集団構造の解明
水圏に生息する生物は、個体や配偶子の行き来が水の中で自由に起きているように見えますが、実際には生活史の違いに応じて様々な程度の隔離が生じています。隔離によって遺伝的に異なる集団が存在するようになることを「集団の構造化」と呼びます。集団構造はDNA分析を通じてとらえることができ、集団間の分岐年代や移住率を推定することが可能です。このことは、個々の集団について過去から現在にいたるまでの進化史を再構築し、隠されていた生態学的特徴を明らかにすることにもつながります。経済的に重要な魚介類はもちろんですが、様々な環境に生息する水圏生物についてデータを集め、個々の生物に応じた保全管理の方策について提言しています。また、個々の地域集団が示す生活史形質の変異についても着目し、DNA分析で得られた集団構造データをフレームワークとして、地域適応の遺伝的基盤を解明する試みもスタートしました。
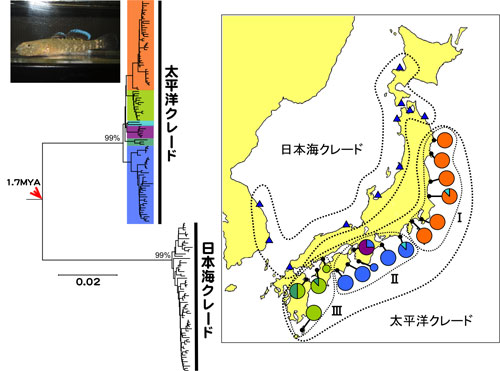
| 図1. | 日本列島沿岸におけるアゴハゼ集団の遺伝的分化.アゴハゼは日本列島の潮間帯で普通に見られますが,そのミトコンドリアDNAの系統は太平洋側と日本海側 とで大きく2つに分かれており,これらは170万年前に分岐したことが明らかにされました(Hirase et al., 2012). |
2)人工種苗の遺伝的特性の把握と放流効果に関する研究
天然水産資源の維持増大を図ることを目的として、人工種苗の放流事業が盛んに行われてきました。資源の増大維持に貢献した事例も確かにありましたが、人工種苗自体の遺伝的劣化や劣化した種苗が天然集団の再生産に寄与することによって起こる遺伝的多様性の変化も懸念されるようになりました。この懸念への回答を得るためには、人工種苗の遺伝的特徴や放流された種苗の天然水域での生残率あるいは再生産への寄与度を調べる必要があります。天然集団との比較から人工種苗の遺伝的特性を浮き彫りにし、天然水域での人工種苗の生態を様々なDNA鑑定手法を用いて明らかにすることによって、より良い放流方法の確立に貢献することを目指しています。
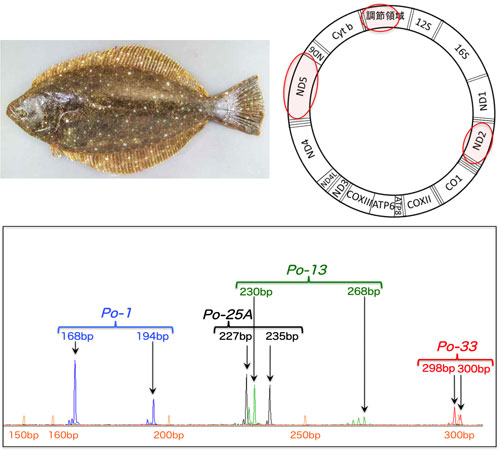
| 図2. | ヒラメ人工種苗のDNA鑑定に使用されるミトコンドリアDNAの遺伝子(右上)とマイクロサテライトDNAのマルチプレックスパネル(下). |
2.アワビやナマコの新品種開発に関する研究
高級食材であるアワビやナマコの養殖を行う上で有用となる形質(高い生残率、速い成長、味など)の遺伝的基盤を明らかにし、養殖に適した品種の開発を目的としています。地域特有の形質が固定された品種を作出できれば、地域ブランドの創出につながるかもしれません。屋内外の飼育施設を用いた交配実験を基軸として、上記の分子生態学的な研究やその他のゲノム情報も活用しながら、世界でも例のないアワビやナマコの新品種を作成および維持することを目指して研究を行っています。
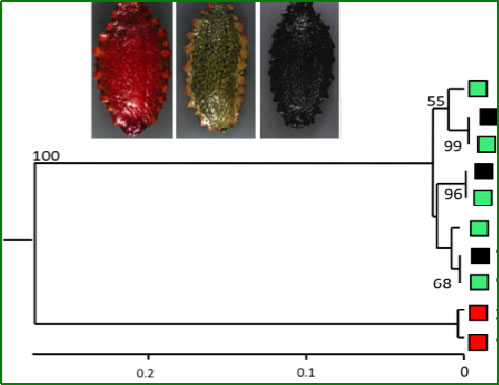
| 図3. | マナマコ色彩変異型間の遺伝的類縁関係.マナマコには、アカ(高級)、アオ(普通)、クロ(安価)の3種類の色彩変異型があります。これらの遺伝的関係を調べた結果、アオ・クロとアカの2グループに分かれました(Kanno et al., 2007)。 この2グループの遺伝的特性をうまくミックスすることで、アカの美味しさとアオ・クロの高い繁殖力を併せ持つナマコの新品種を生み出せるかもしれません。 |
3.その他の研究
その他にも海洋空間の有効利用や生態系の撹乱防止を目指した研究も行われています。一例として、バラスト水中の外来生物除去に関する研究があります。船舶のバラスト水に混入して排出された様々な水生生物が外来種として各地の生態系に影響を与えています。バラスト水そのものを変成させることなく、混入した外来生物だけを効率良く除去するシステムを開発できれば、これ以上の生態系撹乱を防止することができるはずです。民間企業との共同研究によってマイクロ・ナノバブルを利用した効率的除去システムの開発研究を推進しています。

図4. バラスト水中の外来生物を効率的に除去するシステムを開発するために作られた実験装置.
