植物遺伝育種学研究室での
DNA変異解析による育種技術の開発について
(2014.7.1 更新)
育種においては、望ましい遺伝子型の個体や系統を選ぶ「選抜」が最も重要な過程です。しかし、成分特性や耐病性など表現型では選抜しにくい特性があるため、それら特性に関わる遺伝子の望ましい対立遺伝子を持つものがDNA
分析で選抜できれば、育種が効率的に行えるだけでなく、遺伝子情報に基づいたより科学的な育種が可能となります。望ましい対立遺伝子と望ましくない対立遺伝子とは一塩基の違いしかないことがあるため、DNA
分析を育種に利用するには、一塩基多型(Single nucleotide polymorphism, SNP)が簡易に低コストで分析できることが必要です。そのため当研究室では、SNP
の低コスト簡易分析技術の開発を行ってきました。多数あるSNP 分析法の中で、当研究室で開発しましたドットブロットSNP 法は、低コストで一度に多数個体の分析が可能であることから、育種の現場でも利用できるものと期待しています。
1. 未同定SNP を検出するための電気泳動法を利用した方法の改良
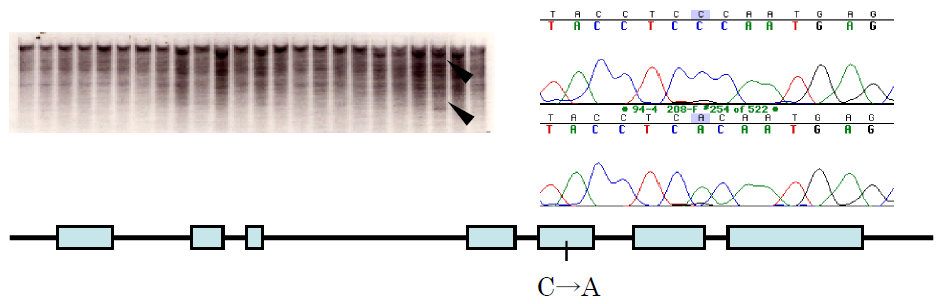
目的とする遺伝子の中のどの塩基に変異(多型)があるか不明な場合は、塩基配列を決定してSNP を同定しますが、塩基配列決定にはコストがかかります。電気泳動法を利用すれば、低コストで多数試料の遺伝子について変異の有無を分析できます。
最初に用いた方法は日本人により開発されたSSCP(single strand conformation polymorphism)法ですが、小さなDNA
断片(500 塩基以下)の変異しか検出できず、断片内での変異の位置によっては差が検出できないことがありました。そこで、PCR(polymerase
chain reaction)で増幅した2,000 塩基対程度のDNA 断片を複数の四塩基認識制限酵素で切断し、SSCP 分析する方法(PCR-RF-SSCP
分析法)に切り替えたところ、効率よくSNP を見つけることが可能になりました。この方法でモチ性突然変異体のwx遺伝子を分析し、その原因となる一塩基多型を数多く見つけ出すことに成功しました
(Sato & Nishio 2003 Theor Appl Genet 107, 560-567)。またこの方法を用いて、イネの多数の品種の遺伝子DNA
変異を見出しました(Shirasawa et al. 2004 DNA Res 11, 275-283)。
セロリから抽出した酵素を用いて二本鎖DNA のミスマッチ部位を切断して電気泳動分析し、SNP がその断片中にあるかどうか分析する方法は、化学変異剤で突然変異誘発した集団からDNA
分析で突然変異体を選抜する方法(TILLING 法と呼ばれる)として利用されています。当研究室では、アブラナ科野菜のミズナから抽出した酵素を用いた一塩基多型分析に改変し、ガンマ線照射により人為的に誘発した突然変異体の逆遺伝学的な選抜が可能になりました(Sato
et al. 2006 Breeding Sci 56, 179-183)。
2. ドットブロットSNP 分析法の開発
電気泳動法を用いた分析では、1 人で1 日300 サンプルの分析が限界で、多数個体や多数遺伝子の分析には不向きです。そこで、多検体の一塩基多型を一度に簡単に判定する技術を開発しました(Shirasawa
et al. 2006 Theor Appl Genet 113, 147-155)。この方法は、PCR で増幅したDNA をナイロン膜に点(ドット)で付け、検出したい塩基配列を持つ標識した17
塩基のオリゴヌクレオチドと非標識の対立遺伝子に結合するオリゴヌクレオチドをドットに結合させて検出する方法です。類似の方法はかなり以前に報告されていましたが、ほとんど使われていなかったもので、本方法はその技術的な問題を解決したものです。

この器械で8 cm×12 cm のナイロン膜に864 サンプルのDNA を載せます。

点(ドット)のシグナルとして一塩基多型(SNP)を判定
ドットブロットSNP 法では、2 枚のナイロン膜で864 個体の遺伝子型分析が可能で、一人で同時に10 枚~20 枚のナイロン膜を扱えますので、一度に4,000~8,000
個体以上の分析が出来、育種現場で扱う個体数の分析が低コストで可能です。多数個体を扱う場合、PCR のための植物DNA の調整に労力がかかりますが、本方法ではPCR
で増幅するDNA 断片が小さくてもよいため、葉からミニコルクボーラーで葉を切り取るだけでDNA サンプルと出来ることも示しました(Shiokai
et al. 2009 Mol Breed 23, 329-336)
本方法は多数個体で少数の遺伝子について遺伝子型分析を行うのに適していますが、多数の遺伝子について分析するには、それぞれのSNP について標識オリゴヌクレオチドを合成しなければならず、コストがかかります。この問題を解決するため、間接標識でドットのシグナルを検出する改変法も開発しました(Shiokai
et al. 2010 Mol Breed 25, 179-186)。また、マーカーごとに最適分析条件を検討しなければなりませんが、多数マーカーを扱う場合には大変手間がかかりますので、オリゴヌクレオチドの塩基配列から最適検出条件を推定する方法も明らかにしました(Shiokai
et al. 2010 Plant Cell Rep 29, 829-834)。
ドットブロットSNP 法やその改良法を用いて、多数の遺伝子のSNP 分析を行い、ダイコンやB. rapa 、B. oleracea の連鎖地図を作成し、イネやダイコン等のストレス耐性や成分特性等のQTL
解析を行いました(「植物遺伝育種学研究室でのゲノムと遺伝資源の研究」、「植物遺伝育種学研究室での環境ストレス耐性の遺伝育種に関する研究」参照)。18
のSNP マーカーで10 ha 以上栽培されている日本のイネ品種の判別がほとんど出来ることも示しました(Sato et al. Breeding
Sci 60, 447-451)。
3. 磁気ビーズを用いたSNP 分析法の開発
ドットブロットSNP 法は、少数個体の分析結果をすぐにその日のうちに出したいときにはあまり適しません。少数個体の多数遺伝子の分析をしたいときにはやや面倒です。また、分析用機器の温度が不正確な場合には、あまり明確な結果が得られません。そのため、より簡便で短時間で出来る方法を開発しました。ドットブロットSNP
法と原理は同じですが、ナイロン膜の代わりにストレプトアビジン被覆磁気ビーズを用いる方法で、PCR 用の機械一台で分析でき、溶液中での反応のため温度条件設定も容易で、その日のうちに結果が出ます。
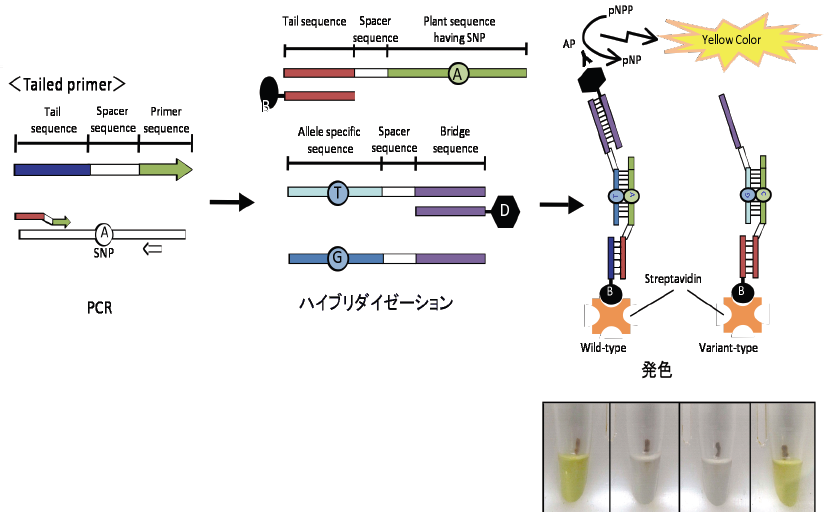
磁気ビーズ法でのSNP 分析の原理と検出されるシグナル(発色)
磁気ビーズを用いた方法の欠点は、磁気ビーズのコストがかかることです。コストの点では、ドットブロットSNP 法にはとてもかないませんが、簡単で便利です。SNP
分析だけでなく、アブラナ類の種判別や、アブラナ類やバラ科果樹のSハプロタイプ同定(Wang et al. 2013 Plant Cell Rep
32, 567-576)にも使えます。
上記以外にも、いろいろやっています。興味のある方は、お気軽にお問い合わせ下さい。お問い合わせ先は、植物遺伝育種学研究室のトップページから。
