アブラナ類のゲノム(その生物の生存に必要な最小限の染色体のセット)の研究は、私たちの研究室で長い伝統があります。ハクサイやカブの学名はBrassica rapa L.、キャベツやブロッコリーはBrassica oleracea L.、セイヨウナタネはBrassica napus L.、カラシナはBrassica juncea Czern.ですが、これらを含むBrassica 属の6 種のゲノムの関係が、花粉母細胞の減数分裂のときの染色体対合の観察から日本で解明され、そのゲノム関係はUの三角形として世界的に知られています。この研究に当研究室の初代教授である水島教授が大きく貢献され、その他のアブラナ近縁種との関係も研究されました(Mizushima 1980 Brassica crops and wild-allies pp89-106)。
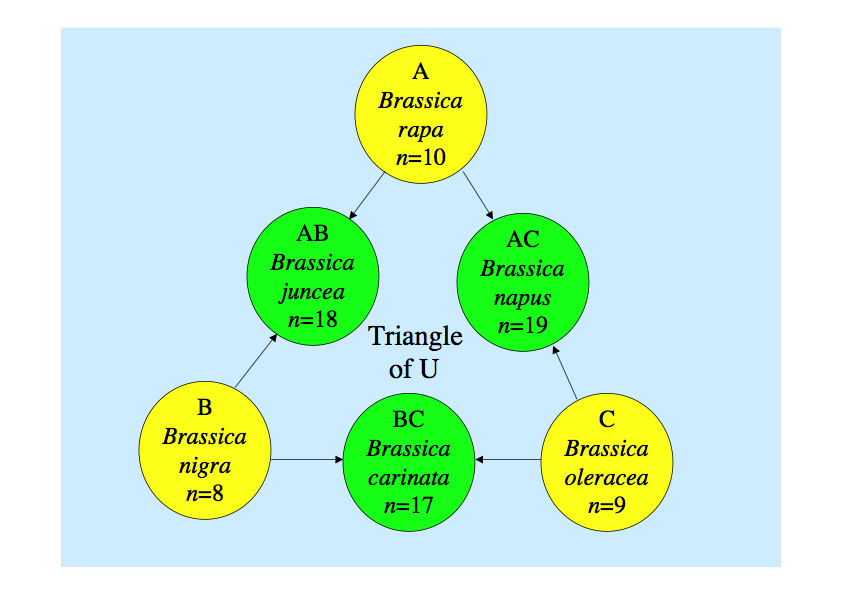
Brassica 属植物のゲノムの関係を示すUの三角形
現在では、ゲノムという用語は、生物が持つ全遺伝情報と言う意味で用いられ、ゲノム研究とは、全染色体のDNA の塩基配列を端から端まで決定することを意味します。ゲノムの塩基配列を決定するためと、アブラナ類の種々の特性の遺伝子を同定するためには、DNA
マーカーを多数作成し、連鎖地図を構築する必要があります。ハクサイ(B. rapa)、キャベツ(B. oleracea)、ダイコン(Raphanus
sativus)の様々な形質に関連する遺伝子を解明することを目的に、多数のDNA マーカーを作成し、連鎖地図を作成しました。特に、dot-blot-
SNP 法(Shirasawa et al. 2006 Theor Appl Genet 113, 147-155 DNA 変異解析による育種技術参照)によるSNP(single
nucleotide polymorphism 一塩基多型)マーカーを多数利用しています。これらのDNA マーカーを使って、グルコシノレート含量(Zou
et al. 2013 Plos One 8, e53541)、開花の早晩性(Li et al. 2009 DNA Res 16, 311-323)、葉毛の変異(Li
et al. 2013 Theor Appl Genet 126, 1227-1236)、耐病性(Kifuji et al. 2013 Euphytica
190, 289-295)などの遺伝的特性に関するQTL(Quantitative Trait Loci 量的遺伝子座)を見出し、これら特性に関わる遺伝子を同定あるいは推定しました。
B. rapa とB. oleracea は縁が近いにも関わらず、染色体数がそれぞれn=10 とn=9 と異なります。遺伝子の連鎖地図の比較により、この違いは、1
本の染色体が切れてn=9 からn=10 となったというような単純なものではなく、進化の過程でいくつかの染色体が切れたりつながったりして複雑な染色体構造の変化が起こったことによるものであることが分かります。しかし、第1
染色体と第2 染色体は2 種で一致しており、B. rapa の第7 染色体はB. oleracea の第6 染色体と、B. rapaの第9 染色体はB.
oleracea の第8 染色体と一致していることが分かります(Ashutosh et al. 2012 Mol Breed 30, 1781-1792)。B.
rapa とダイコン(R. sativus)の間では、より複雑な関係になっていることが分かり(Li et al. 2011 DNA Res 18,
401-411)、ダイコンの方がB. oleracea よりもB. rapaから遠縁であることを反映しています。かつて減数分裂時の染色体の対合で染色体の相同性を推定していたゲノム研究が、分子レベルでより詳細にできるようになりました。全ゲノムの塩基配列が分かれば、この関係がより厳密に分かるようになり、アブラナ類のゲノム進化の課程が推定できるようになります。
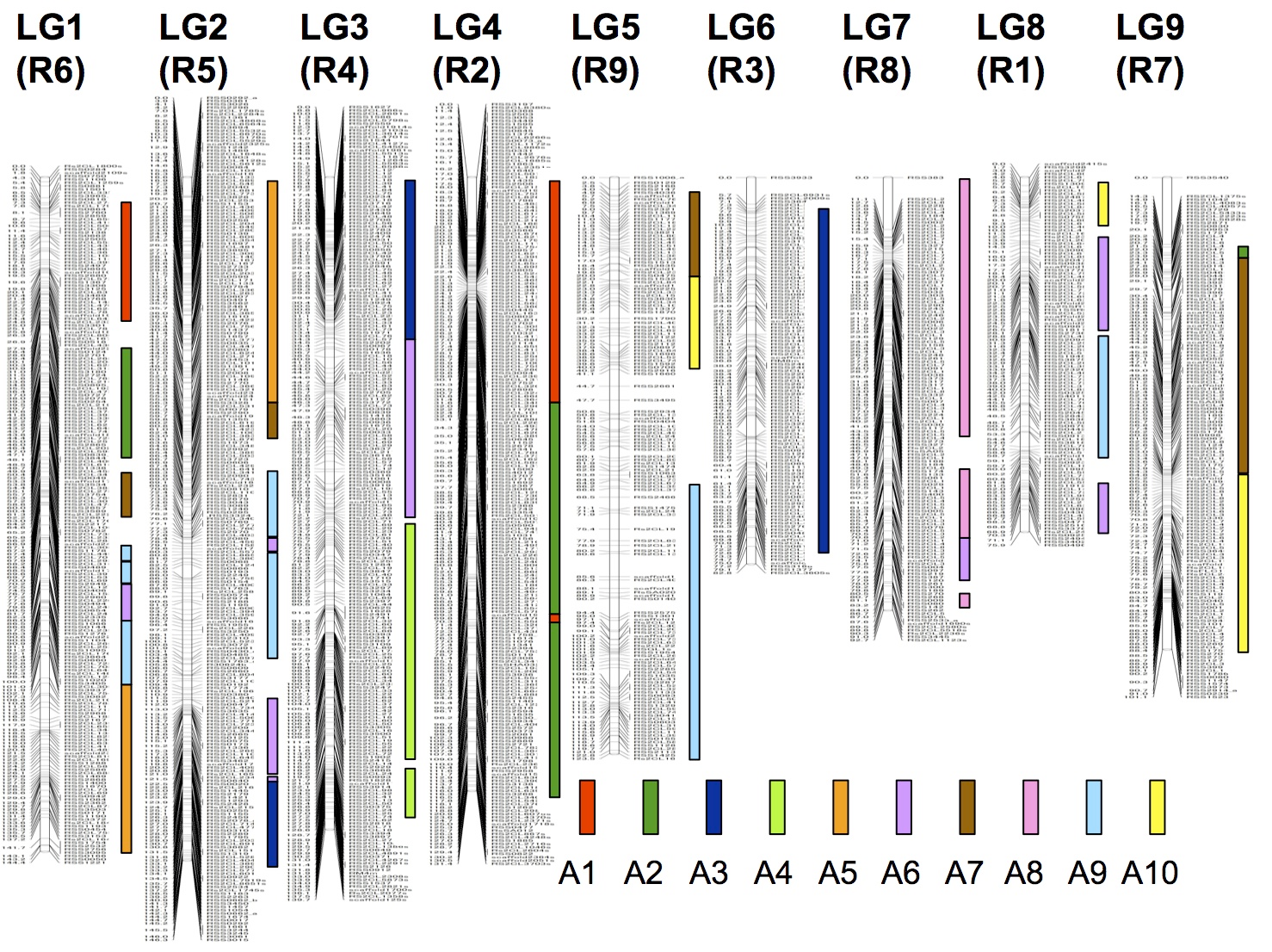
ダイコンの遺伝子連鎖地図のBrassica rapa との対応(右横のバー)
2008 年より、生研センターの研究費支援を受けて、ダイコンゲノム研究を行いました。ゲノム塩基配列を決定するため、当研究室で作成していたSNP
マーカーの連鎖地図とかずさDNA 研究所で作成された連鎖地図と統合し、さらにSNP マーカーを加えて、約2,550 からなる高密度連鎖地図を作成しました。次世代シーケンサーでゲノム塩基配列データを多数得て、BAC(Bacterial
Artificial Chromosome)クローンの末端塩基配列も決定し、かずさDNA 研究所の協力を得てそれらのデータを統合し、ダイコンゲノムの76%にあたる約400
Mb(4 億塩基対)の長さの塩基配列を世界に先駆けて決定しました。これらのゲノム塩基配列情報は、‘Raphanus sativus Genome
Data Base’ (http://radish.kazusa.or.jp/) で公開しました。また、岩手大学、野菜茶業研究所、タキイ種苗株式会社と協力して、8 品種間の1,000 以上の遺伝子について塩基配列変異を明らかにし、育種に有用な情報を得ました(Kitashiba
et al. 2014 DNA Res doi:10.1093)。
本研究室ではアブラナ類の遺伝育種の研究に伝統があるため、アブラナを使った遺伝学的研究や育種にとって重要となるアブラナ遺伝資源を多数保存しており、それらの種子を世界の研究者の要望に応じて提供しています(Tohoku
Univ. Brassica Seed Bank 参照)。保有するアブラナ遺伝資源の中には、専門家の間でもほとんど名が知られていない種が多数あることから、種名の混乱を避けるため、アブラナ近縁種の種同定技術を開発しました(Tonosaki
and Nishio 2010 Plant Cell Rep 29, 1179-1186, Tonosaki et al. 2013 Mol
Breed 31, 419-428)。また、アブラナ遺伝資源の有効な利用を図るため、それらの類縁関係を遺伝子の塩基配列分析により明らかにしています
(Inaba and Nishio 2002 Theor Appl Genet 105, 1159-1165)。このアブラナ遺伝資源は希少価値の高いもので、世界的に知られています(→Tohoku Univ. Brassica Seed Bankへ)。
上記以外にも、いろいろやっています。興味のある方は、お気軽にお問い合わせ下さい。お問い合わせ先は、植物遺伝育種学研究室のトップページから。
